|
Resumen Bolivia es un país esencialmente forestal, ya que alrededor del 48 % de su territorio esta cubierto por bosque. Si bien se han hecho grandes avances en la estrategia de aprovechamiento sostenible de los recursos forestales, sin embargo aún resta mucho que hacer, especialmente en lo que concierne a la aplicación de las biotecnologías. Una especie estratégica de los andes es la tara (Caesalpinia spinosa Molina Kuntze), especie que se encuentra ampliamente distribuida a lo largo del territorio boliviano, principalmente en los departamentos de Cochabamba, Santa Cruz, Chuquisaca, Potosí, La Paz y Tarija entre los 1000 y 3000 msnm. A partir de árboles georeferenciados y la aplicación del SIC se logró un mapa de su distribución en la región andina especialmente en Perú, donde se aprovechan los frutos para su exportación a un mercado internacional de alta demanda. A fin de indagar la variabilidad respecto a su distribución, se caracteriza mediante el empleo de marcadores moleculares (RAPDs), midiéndose el polimorfismo de las bandas, los cuales presentaron un 70 % de similitud. La interpretación de estas bandas permite inferir que las dos procedencias de la tara analizadas son similares, revelando que se puede tratar de la misma variedad, siendo necesario realizar confirmación de estos resultados con un nuevo análisis incrementando el universo de las muestras, considerando además otras procedencias en el análisis y el uso de otros marcadores moleculares. Introducción Bolivia esta declarado como un país m con incontables especies. Esta condición, constituye una fuente de estudio interminable de sus recursos, especialmente forestales. Se dice que a nivel mundial menos del 10 % de la flora ha sido estudiada químicamente, en este contexto una especie que actualmente despierta interés es la tara (Caesalpinia spinosa Molina Kuntze). La vaina de la tara es usada como materia prima para la obtención del ácido tánico, muy usado en las industrias del cuero de alta calidad, farmacéutica, química, pinturas, entre otras. De la semilla se obtiene una goma de uso alimenticio, alternativa a las gomas tradicionales en la industria mundial de alimentos para consumo humano. La tara se distribuye en los valles de la región andina, éste recurso forestal es producido en varias zonas del país, siendo sus principales sitios de producción los valles de los departamentos de Cochabamba, Santa Cruz, Chuquisaca, Potosí, La Paz y Tarija. El mejoramiento genético y su caracterización varietal, es importante para poder determinar si el contenido de tanino que presenta la tara, en las diferentes regiones del país, esta ligado al ambiente y condiciones de suelo o es una característica genética. De esta forma el presente trabajo constituye un documento indicativo de esta posible condición, realizado con dos procedencias de tara (Santa Cruz y Cochabamba). El objetivo del estudio fue realizar un mapeo de la distribución de la especie y la caracterización molecular preliminar de las dos procedencias de tara, antes señaladas, mediante la utilización de marcadores moleculares. Revisión de literatura El SIG y distribución geográfica. La aplicación del Sistema de Información Geográfica (SIG) mediante el Florama, se constituye en una herramienta que permite determinar la distribución de los recursos genéticos a distintas probabilidades de ocurrencia, cuyo producto final es un mapa de alta precisión (Jones y Gladkov, 2003). Identidad genética Marcadores moleculares. De manera general, un marcador puede definirse como cualquier característica (morfológica, bioquímica, molecular) que permite revelar diferencias entre individuos. Según la dimensión en la cual los marcadores son revelados, se les puede clasificar en tres tipos: Marcadores Morfológicos, Bioquímicos y Moleculares (Rojas, 2005). Según Rojas (2004), un marcador molecular es una característica que tiene que estar esencialmente determinada por la naturaleza de los genes que la influencian y el polimorfismo. Se clasifican según la técnica que se emplea:
Tipos de marcadores moleculares. Existe un sin número de marcadores que se vienen empleando en la diferenciación de individuos, discriminación entre clones, análisis filogenéticos y taxonómicos, dispersión de especies entre otros. Los RAPDs (Random Amplifled Polymorphic DNAs). La técnica RAPDs, es una de las técnicas más versátiles, consiste en la amplificación mediante PCR de un ADN anónimo, utilizando para ello un cebador de secuencia arbitraria. Normalmente se usa un solo cebador, de 10 nucleótidos de longitud y con un contenido alto en G+C, pe. OP-A01. Posteriormente, se realiza la amplificación de ADN anónimo con cebadores de secuencia arbitraria y la correspondencia entre fragmentos amplificados y bandas en un gel, obteniéndose los RAPDs. El procedimiento básico para RAPDs, se detalla a continuación:
Existen diferentes métodos de separación / detección. Como ejemplo se tiene: Agarosa/bromuro de etidio y Poliacrilamida/nitrato de plata. La técnica es cómoda, rápida, requiere poco ADN, no presupone conocimiento previo sobre el genoma analizado y se puede distinguir rápida y simultáneamente muchos organismos. El principal inconveniente de la RAPDs es que es una técnica poco reproducible. Interpretación de bandas de RAPDs. La interpretación de bandas es bastante importante, de esta manera se consideran los siguientes aspectos: Subjetividad: ¿Qué bandas se incluyen en el análisis y cuáles no? Problemas de la co-migración: - Una banda del mismo tamaño en dos individuos NO representa necesariamente el mismo fragmento de ADN. - Una banda NO contiene necesariamente un único fragmento de ADN (Rojas, 2004). Materiales y métodos Material para la determinación de la distribución geográfica: GPS, Programa FloraMap. Material para la determinación de la identidad genética: ● Material vegetativo: Hojas tiernas de tara procedentes de Santa Cruz (Valle Grande) y Cochabamba (Tarata). ● Materiales para la extracción de ADN, PCR, Kits (marcadores moleculares), materiales para la electroforesis, Secuenciador, entre otros. La metodología empleada se puede resumir en los siguientes puntos: Distribución geográfica El Centro de Semillas Forestales, con el apoyo del Programa del Manejo del Recurso Forestal Tara y el Sistema Nacional de Conservación de Recursos Genéticos (SINARGEAA), realizó la recolección de germoplasma y la georeferenciación con GPS de 100 accesiones de tara distribuidas en toda Bolivia, la información fue introducida y procesada en el programa FloraMap, dando como resultado un mapa de distribución de 50 a 90 % de probabilidad de ocurrencia de la especie en la región andina. Identidad genética La metodología empleada para la determinación de la identidad genética, fue la desarrollada por el laboratorio de Mejoramiento Genético del CRA-W. En líneas generales se consideran los siguientes pasos:
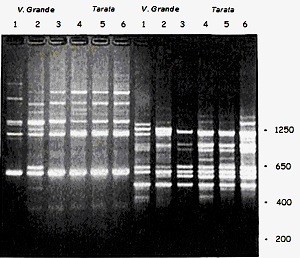
Resultados y discusión Distribución regional de la especie En Bolivia, la especie arbórea se encuentra en los valles, está presente en el departamento de Cochabamba (Tarata, Cliza, Punata, Arani, Sacaba, Sipe Sipe, Vinto, Cercado, Tapacarí, Santivañez) y en algunas regiones montañosas secas, La Paz (Murillo, Sur Yungas), Santa Cruz (Valle Grande, Saipina, Comarapa), Chuquisaca (Sucre, Chaco, Yotala), Potosí (San Pedro de Buena Vista) y Tarija. En el mapa de la portada de esta Revista, se presenta la distribución de la especies en la región (P 0.5-0.95), partiendo de la georeferenciación de 100 accesiones recolectadas por el BASFOR, observándose la presencia de la especie a más de 70 % de probabilidad en el norte argentino, Perú y Ecuador. Determinación de la identidad genética El ensayo preliminar se realizó mediante la comparación de bandas entre las dos procedencias, para la determinación de la identidad genética de las posibles variedades, obteniéndose los siguientes resultados: ● Los marcadores moleculares empleados fueron los RAPDs y midieron el polimorfismo de las bandas los cuales presentaron un 70 % de similitud.
● La interpretación de estas bandas permite inferir que las dos procedencias de la tara analizadas son similares, revelando que se puede tratar de la misma variedad, siendo necesario realizar una confirmación de estos resultados, mediante un nuevo análisis, incrementando el universo de las muestras, considerando otras procedencias en el análisis y el uso de otros marcadores moleculares. ● Este ensayo es considerado preliminar por haberse realizado con un número de muestras bajo, por lo que se consideraron pocas bandas. Además fue necesario poner a punto la extracción de ADN por tratarse de una especie nativa, en la que no se realizaron ensayos moleculares anteriores. ● Otro punto a destacar es que no se cuenta con primers específicos a la especie, lo que dificulta hacer una caracterización molecular mas precisa. Conclusiones ● La aplicación del FloraMap como un programa del SIG, permitió determinar la distribución de la tara en la región andina a diferentes probabilidades, a partir de 100 datos georeferenciados en Bolivia. ● Las dos procedencias analizadas mediante el uso de marcadores moleculares (RAPDs), muestran un 70 % de probabilidad de que se trate de la misma variedad, según la interpretación del polimorfismo de las bandas. ● Es necesario, hacer ensayos complementarios para poder asumir con mayor precisión la identidad genética de las diferentes procedencias de tara de Bolivia. Referencias citadas y sugeridas ALEMÁN, F., UGARTE, C. 2005. Informe de Manejo del Recurso Genético Forestal Tara. BASFOR/ESFOR - FOSEFOR/IC/COSUDE. DRUART, Ph., UGARTE, C. 2006. Caracterización molecular preliminar de tara (Caesalpinia spinosa Molina Kuntze), mediante el uso de marcadores moleculares. BASFOR/ESFOR FOSEFOR/SINARGEAA. FOSEFOR, 2001 Contribución a la fenología de especies nativas forestales de Bolivia y Ecuador. Quito, Ecuador. pp. 50 - 52. JONES, P., GLADKOV, Á. 2003. Flora-Map. CIAT CD - ROM. Series. Colombia. ROJAS. J. 2004. Selección asistida por marcadores moleculares (SAM). Curso de Marcadores Moleculares. PROINPA. ROJAS, J. 2005. Estrategia para la aplicación de tecnologías derivadas de la biología molecular en el sector forestal. BASFOR/ESFOR - UMSS/SINARGEAA-MACA. |